Structure de l'ARN - Définition
La liste des auteurs de cet article est disponible ici.
Structure tertiaire
En plus de leur structure secondaire, constituée d'appariements de base standard, les ARN peuvent adopter une structure tridimensionnelle compacte, bien définie, qui résulte d'interactions additionnelles entre les éléments classiques de structure secondaire (hélices, boucles). Ces interactions additionnelles dans la plupart des cas de type non-canonique, c'est-à-dire qu'il ne s'agit ni d'appariement Watson-Crick, ni d'appariements bancals, de type G-U.
Les appariements non-canoniques et les interactions additionnelles à longues distance sont relativement bien connus, en particulier grâce à la résolution de la structure tridimensionnelle du ribosome, qui en a mis en lumière un très grand nombre. L'utilisation de méthodes phylogénétiques (voir ci-dessus) permet également de repérer certaines de ces interactions non-canoniques, lorsqu'on dispose de la séquence du même ARN chez plusieurs espèce. Il est ainsi possible de rationaliser en partie la manière dont se construit la structure tertiaire des ARN et de réaliser des prédictions de repliement relativement fiables, dont certaines ont pu être validées a posteriori par l'expérience.
Appariements non-canoniques
Les appariements non canoniques sont des interactions entre nucléotides, distincts des d'appariements de base classiques, de type Watson-Crick (A-U et G-C) ou bancals (wobble, G-U). On a observé une grande variété de ces appariements dans les structures tridimensionnelles d'ARN résolues par cristallographie ou par RMN. Ces appariements non-canoniques impliquent toujours des liaisons hydrogènes entre les bases, qui sont coplanaires, comme dans les paires Watson-Crick.
Une nomenclature systématique de toutes ces interactions a été proposée par Eric Westhof et ses collaborateurs. Plus de 150 types d'appariements ont été observés et ont été regroupés en douze grandes familles.
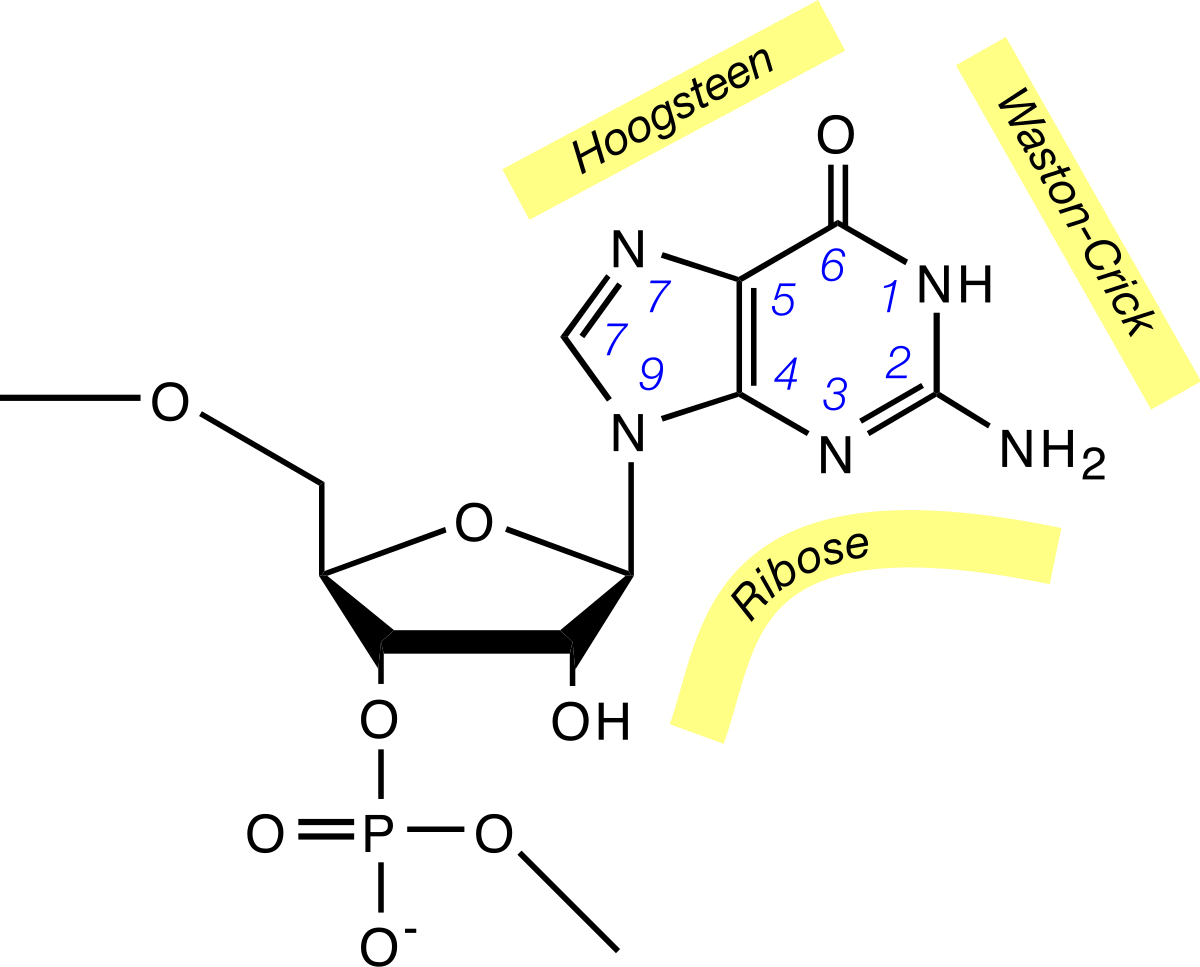
Cette classification repose entre autres sur la face des bases impliquée dans l'interaction. Un nucléotide de type purine comporte par exemple trois faces permettant des liaisons hydrogènes :
- La face Watson-Crick. C'est le côté comportant le N1 et les fonctions oxo et amino en position 2 et 6. C'est cette face qui est impliquée dans les appariements canoniques A-U et G-C, ainsi que dans les appariements bancals G-U
- La face Hoogsteen. C'est le côté comprenant le N7 et la fonction oxo ou amino en position 6.
- La face ribose. C'est le côté comprenant le N3 et la fonction en position 2. La fonction 2' hydroxyle du ribose peut également participer aux interactions sur cette face.
A cette nomenclature d'interactions entre bases, il faut ajouter la possibilité pour cette dernière d'adopter différentes conformations par rapport au ribose auquel elle est liée. La liaison glycosidique qui relie la base au sucre a en effet deux orientations favorables principales, suivant que la face Watson-Crick de la base est située du côté du ribose (orientation cis) ou du côté opposé (orientation trans). Dans les appariements canoniques, les nucléotides sont toujours en conformation trans. Cette orientation du sucre par rapport à la base détermine en particulier la polarité, parallèle ou antiparallèle, des brins d'ARN qui sont appariés.
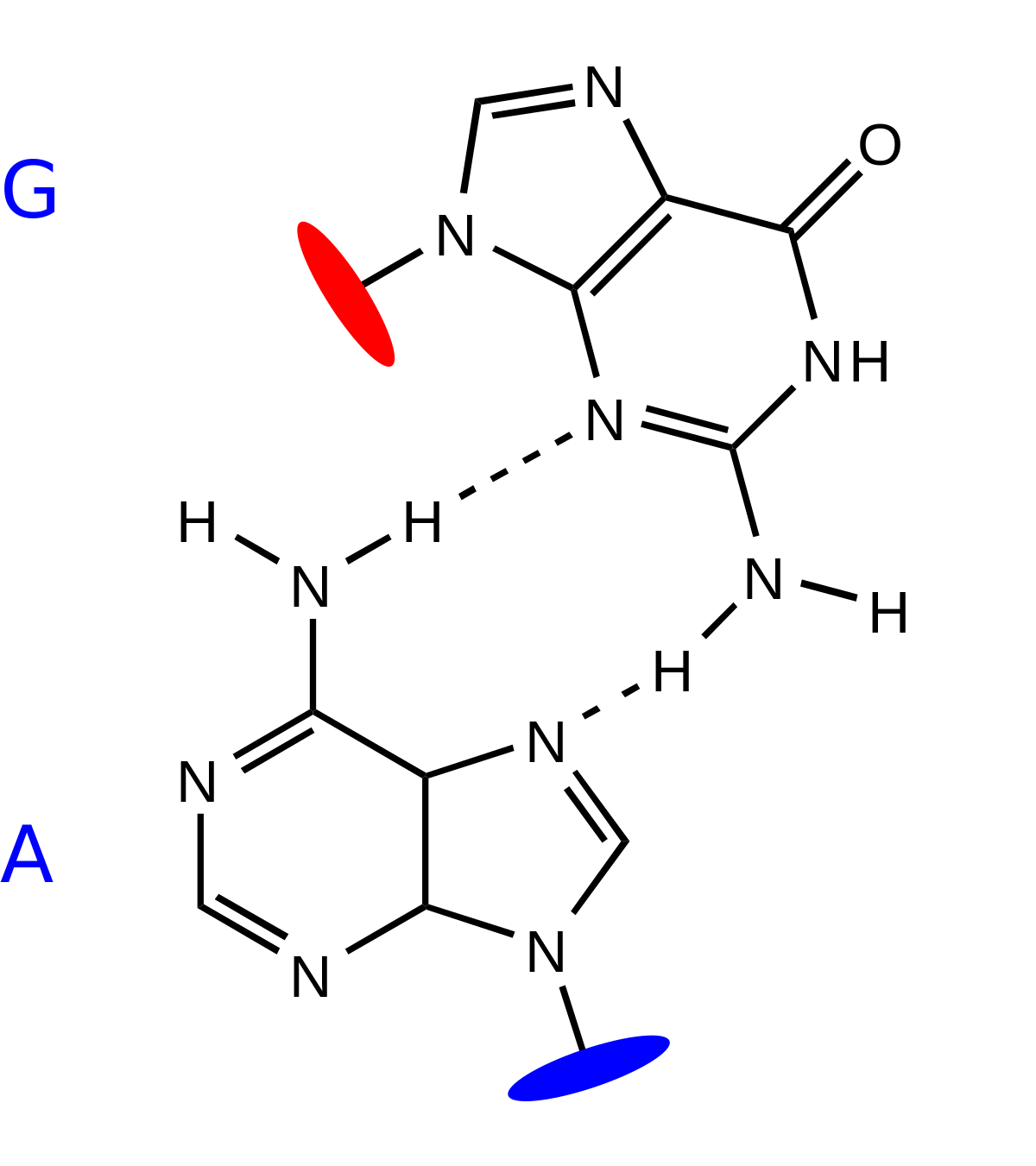
Parmi ces appariements non-canoniques relativement fréquents, on trouve par exemple des appariements Hoogsteen A-U impliquant la face Hoogsteen d'une adénosine et un uracile, des paires G-A "en cisaille" (sheared), impliquant la face Hoogsteen du A et la face ribose du G.
Enfin, on trouve plus rarement des appariements qui impliquent des formes protonées de certaines bases. Ainsi, existe des paires C-C+, où la deuxième cytosine est protonée sur sa position N3. Ce type de paires de bases non-canoniques porte une charge positive et est stabilisé à pH acide (le pKa du N3 de la cytidine est d'environ 4.2).
Motifs récurrents
Tétraboucles
L'analyse de la structure des ARN ribosomiques a permis de mettre en évidence la présence d'un nombre très élevé de structure en hélices fermées par des boucles terminales de quatre nucléotides. Ces boucles, appelées tétraboucles (tetraloop), ont des structures très stables, renforcées par des interactions non-canoniques. On en a identifié plusieurs classes, dont les principales se regroupent dans trois familles :
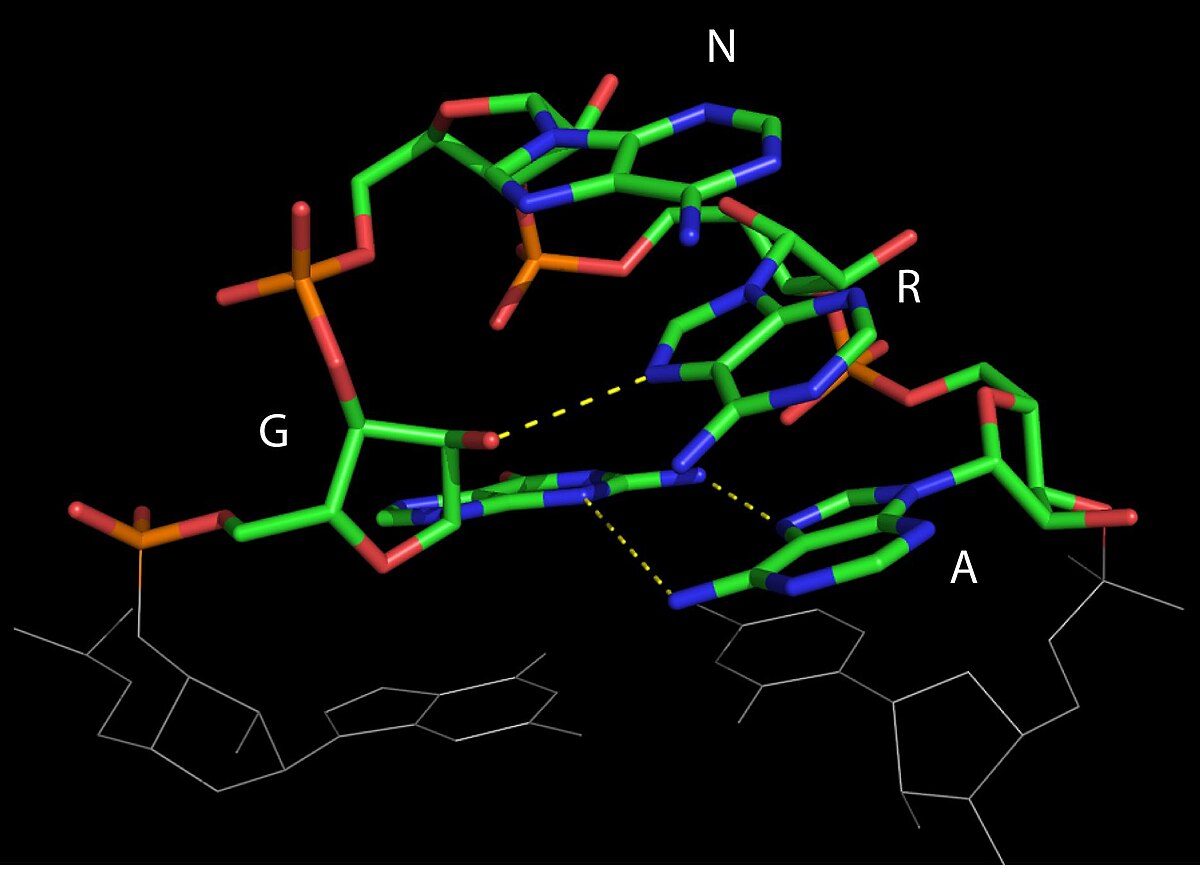
- Les boucles de type GNRA, ou N peut être A, G, C ou U, et R, A ou G. Dans ces boucles (figure ci-contre). Le G en 5' et le A en 3' forment une paire G-A en cisaille. La purine en position 3 est empilée sur l'adénine et fait une liaison avec le 2'-OH du ribose de l'adénine.
- Les boucles de type UNCG, ou N peut être A, G, C ou U.
- Les boucles de type CUUG.
Les tétraboucles de la famille GNRA, qui sont parmi les motifs les plus fréquents dans l'ARN, peuvent former des interactions à longue distance avec des régions en hélice ou en boucle interne. Ces interactions impliquent les deux nucléotides au centre de la boucle qui sont exposés. Ainsi, par exemple, les tétraboucles de séquence GAAA peuvent interagir spécifiquement dans le petit sillon avec des hélices contenant deux paires G-C consécutives et en particulier avec le motif de 11 nucléotides [CCUAAG...UAUGG] où les deux premiers C sont appariés avec deux derniers G. On appelle ces séquences des récepteurs de tétraboucle. Ces interactions tétraboucle-récepteur sont très fréquentes dans les structures tridimensionnelles d'ARN et jouent un rôle important dans le maintien du repliement global.
Boucle E
Interactions à longue distance
Pseudonœuds
Un pseudonœud est une structure formée par l'interaction d'une boucle avec une région située à l'extérieur de la tige qui la délimite. Les structures secondaires canoniques, décrites ci-dessus excluent en principe la formation de pseudonœuds, en particulier parce que leur prédiction par des méthodes bio-informatiques reste assez complexe.
Les pseudonœuds permettent de former des interactions à longue distance, entre deux régions non-appariées dans la structure secondaire. Ce sont donc des éléments importants de la structure tridimensionnelle des ARN. Par rapport aux structures secondaires canoniques, ils sont en général peu nombreux et de dimension limitée dans les ARN structurés, mais ils jouent souvent un rôle important dans la structure et la fonction. La limite sur leur taille a une origine topologique, liée à la formation de l'hélice additionnelle du pseudonœud : celle-ci ne peut en principe pas dépasser un tour d'hélice, soit une dizaine de paires de bases, sinon cela conduirait à un enlacement complet des brins créant un nœud véritable.
Interactions triples
Une interaction triple, ou triplet de bases, est une interaction entre trois bases situées dans le même plan et formant des liaisons hydrogènes entre elles. Elles peuvent être isolées ou se produire de manière consécutive, les triplets formant alors des empilements, comme les paires de bases dans un duplex. On a alors une structure associant localement trois brins d'ARN. Le type le plus fréquent d'interactions triples s'observe au sein d'une hélice classique, lorsque l'un des deux brins contient plusieurs purines consécutives. Un troisième brin d'ADN peut alors former des appariements Hoogsteen avec le brin purique, en s'insérant dans le grand sillon de l'hélice.