Réaction en chaîne par polymérase - Définition
La liste des auteurs de cet article est disponible ici.
Cinétique mesurable d'une PCR
Ce chapitre traite des cinétiques mesurables d'une PCR, car des limitations technologiques rendent inaccessibles les cycles précoces, et généralement une large portion de la partie exponentielle. Son profil apparent (mesurable) adopte plusieurs phases distinctes, plus ou moins développées en fonction de choix méthodologiques.
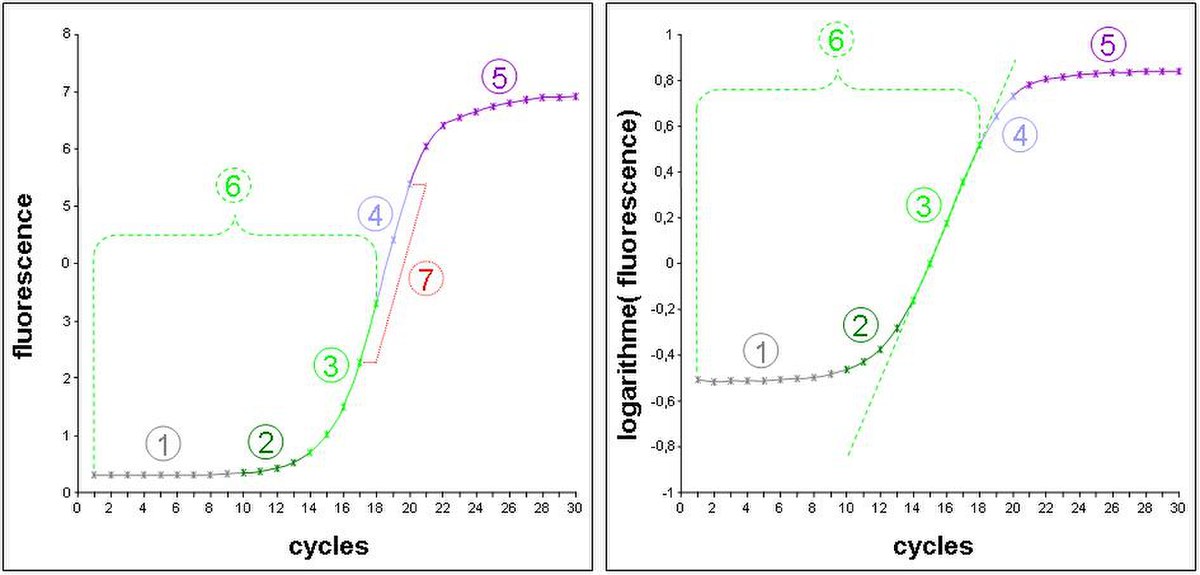
- Schéma de gauche : données expérimentales d’une cinétique de PCR mesurée sur un thermocycleur en temps réel. La fluorescence émise par un intercalant de l’ADN est proportionnelle à la quantité d’ADN présent (c'est-à-dire majoritairement l’amplicon). La courbe représente donc la cinétique d’amplification de l’ADN cycle par cycle.
- Schéma de droite : les valeurs de fluorescence précédentes ont été converties en logarithme décimal. Une cinétique de type exponentiel devrait donc être transformée en un segment de droite (segment quantifiable).
- 1) Bruit de fond de la fluorescence aspécifique du marqueur. Son niveau est très dépendant de la nature du marqueur et du soluté de l’échantillon.
- 2) Zone où la mesure de fluorescence est biaisée par le bruit de fond. Elle se distingue aisément sur le schéma de droite car elle adopte un profil d’exponentielle, alors que les valeurs sont déjà dans un repère semi-logarithmique.
- 3) Zone de la cinétique en phase exponentielle et mesurable sans biais. Elle adopte dans le schéma de gauche l’apparence d’un segment de droite nommé segment quantifiable. Remarquez que dans cet exemple expérimental, il ne fait que 5 cycles.
- 4) Zone de la cinétique où la polymérase devient un facteur limitant. L’accumulation d’amplicon se fait alors de manière constante et devient linéaire sur le schéma de gauche.
- 5) Phases d’amortissement puis de plateau de la cinétique dues à l’apparition de facteurs limitants non constants (dNTP, fluorophore, etc.), de dégradation de l’activité enzymatique, de la qualité de fluorescence…
- 6) Phase de la cinétique qui répondait réellement à une loi exponentielle, mais seule la zone 3 est réellement exploitable (sauf pour le maxima de dérivé seconde en PCR en temps réel).
- 7) Zone linéaire sur le schéma de gauche. Une erreur classique en PCR en point final était de déterminer cette zone grâce à une gamme de cycles pour tenter de faire une mesure quantitative de l’ADNc initial à ce niveau.
Principe
La PCR est une technique basée sur une répétition de cycles de transition de température. Sauf pour certaines méthodologies (par exemple l’utilisation de sondes d’hydrolyse), chaque cycle contient trois étapes détaillées ci-dessous. Par souci de didactisme, nous allons considérer pour l’exemple qui suit une efficacité de PCR de 100%.
Les différentes étapes de la PCR
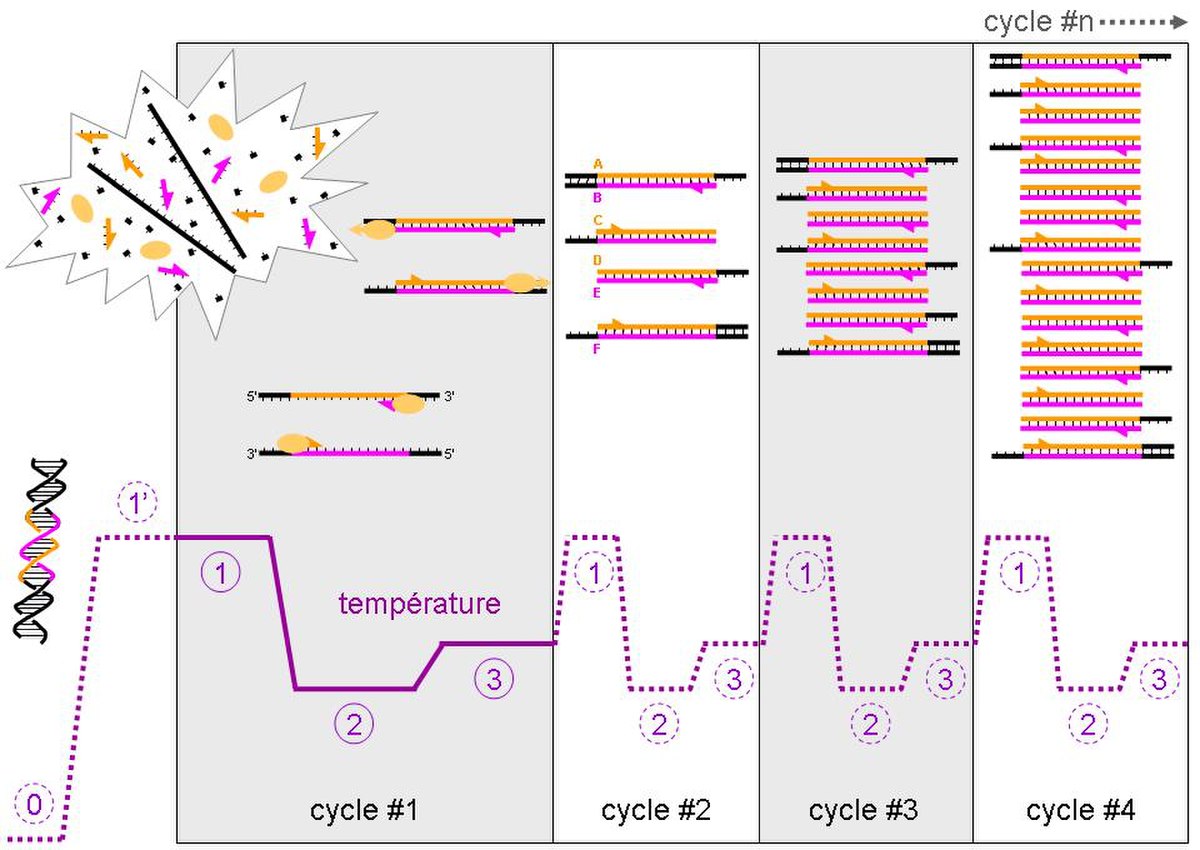
Conditions natives (0 sur le schéma)
Cette étape se fait généralement à température ambiante. L’ADN bicaténaire adopte sa conformation en double hélice. Dans cet exemple, nous considérerons qu’il n’y a qu’une molécule initiale d’ADN double brin dans la solution, la zone colorée (rose et orange) correspondant à notre amplicon. Note: les ADN complémentaires issus de la transcription inverse sont généralement simple brin et adopteront alors de complexes conformations tridimensionnelles similaires à celle des ARN.
Dénaturation initiale (1’ sur le schéma)
Avant de commencer les cycles de PCR proprement dit, une étape de chauffage (généralement 10 à 15 minutes à 95°C) est réalisée. Cette étape permet de : déshybrider les ADN double brin, de casser les structures secondaires, d’homogénéiser le milieu réactionnel par agitation thermique, d’activer les polymérases de type « Hot start », de dénaturer d’autres enzymes qui pourraient être dans la solution (Transcriptase Inverse, Uracil-N-Glycosylase).
Phase de dénaturation (1 sur le schéma)
Cette étape (généralement 0 à 1 minute à 95°C) permet de déshybrider les ADN, de « décrocher » les polymérases qui seraient encore liées à une matrice et d’homogénéiser le milieu réactionnel.
Phase d’hybridation ou d'appariement des amorces (2 sur le schéma)
Cette étape (généralement 2 à 60 secondes à 56-64°C) permet aux amorces sens et anti-sens de s’hybrider aux ADN matrice grâce à une température qui leur est thermodynamiquement favorable. Peu de brins d’ADN matrice peuvent s’hybrider (se lier) avec leur brin complémentaire, ce qui empêcherait la fixation des amorces, car ces dernières sont bien plus courtes et en concentration bien plus importante. Expérimentalement, il est constaté que la PCR fonctionne même avec une phase d’hybridation avec une température supérieure de quelques degrés au Tm théorique des amorces, probablement parce qu’elles interagissent déjà avec les polymérases, qui stabiliseraient leur hybridation à l’ADN matrice.
Caractéristiques des amorces
- les séquences nucléotidiques des amorces doivent être spécifique des séquences complémentaires de l'ADN simple-brin auxquelles elles vont s'apparier. La complémentarité parfaite n'étant par ailleurs pas obligatoire. De plus la spécificité de la séquence est importante dans le sens ou celle-ci ne doit pas pouvoir s'apparier à une autre séquence de l'ADN que l'on souhaite répliquer.
- Les séquences des amorces doivent être choisie en sorte de minimiser les possibilités d'appariement entre elles. De même chaque amorces est choisie pour ne pas pouvoir former une structure secondaire.
- Le procédé même de la PCR reposant, entre autres, sur des équilibres thermodynamiques, les amorces doivent avoir des températures de fusion le plus proche possible, autrement dit le rapport entre les bases AT et GC des deux amorces ne doit pas être trop différent.
Phase d’élongation (3 sur le schéma)
Cette étape (généralement 4 à 120 secondes à 72°C) permet aux polymérases de synthétiser le brin complémentaire de leur ADN matrice à une température qui leur est optimale. Ce brin est fabriqué à partir des dNTPs libres présents dans le milieu réactionnel. La durée de cette étape dépend normalement de la longueur de l’amplicon.
Evolution de l'ADN au cours des 4 premiers cycles
Cycle n°1
-
- Lors de la phase 1, nous constatons que l’ADN initial a adopté une conformation « linéaire » (sans structure secondaire) et simple brin. Les amorces, les dNTPs et les polymérases sont en large excès et répartis de façon homogène dans la solution.
- Lors de la phase 2, une des amorces sens s’hybride avec sa séquence complémentaire sur le brin anti-sens (en rose), une des amorces anti-sens se liant elle au brin sens (en orange). Deux polymérases peuvent alors interagir avec les deux complexes amorces/ADN matrice.
- Lors de la phase 3, les polymérases parcourent leur brin matrice de son extrémité 3’ vers son extrémité 5’ tout en synthétisant le brin complémentaire. Elles s’arrêteront à la fin du cycle, décrochées par la phase de dénaturation du cycle suivant. Les ADN néo-synthétisés sont donc précisément définis à leur extrémité 5’ mais pas à leur extrémités 3’ (parties noires). Les ADN sont alors bicaténaires sur une longueur plus ou moins importante.
- A la fin de l’étape 3, nous avons alors deux brins d’ADN matrice et deux brins (un sens et un anti-sens) d’ADN précisément définis à leur extrémité 5’ uniquement.
Cycle n°2
Les trois phases se déroulent de la même manière qu’au cycle no 1, sauf que deux polymérases arrivées au bout de leur ADN matrice se décrochent spontanément. À la fin de la phase 3, nous obtenons tous les types d’ADN qui existeront lors de la PCR, soit :
-
- Un brin d’ADN natif sens (A).
- Deux brins d’ADN sens précisément définis à leur extrémité 5’ uniquement (B).
- Un brin d’ADN anti-sens correspondant à l’amplicon, c'est-à-dire précisément défini à ses deux extrémités (C).
- Deux brins d’ADN anti-sens précisément définis à leur extrémité 5’ uniquement (D).
- Un brin d’ADN sens correspondant à l’amplicon, c'est-à-dire précisément défini à ses deux extrémités (E).
- Un brin d’ADN natif anti-sens (F).
Cycle n°3
Idem au cycle 2. À la fin de la phase 3, nous observons 1 brin de type A et F, 3 de B et D, et 4 de C et E. Nous observons l’apparition de deux molécules d’ADN double brins C-E qui correspond à notre amplicon.
Cycle n°4
Idem au cycle 3. À la fin de la phase 4, nous observons 1 brin de type A et F, 4 de B et D, et 11 de C et E. Nous observons que l’amplicon devient la combinaison majoritaire.
Cycles au-delà de 4
Si nous avions augmenté le nombre de cycles, nous aurions obtenu le tableau suivant :
| cycle numéro : | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | n | ||
| A | = An − 1 + 0 | |||||||||||||||||
| B | = Bn − 1 + 1 | |||||||||||||||||
| C | = Cn − 1 + (Bn − 1 + En − 1) | |||||||||||||||||
| D | = Dn − 1 + 1 | |||||||||||||||||
| E | = En − 1 + (Cn − 1 + Dn − 1) | |||||||||||||||||
| F | = Fn − 1 + 0 | |||||||||||||||||
| = Σ An à Fn | ||||||||||||||||||
| = 100 * (Cn + En) / Σsimplebrin | ||||||||||||||||||
| = Σsimplebrin / 2 | ||||||||||||||||||
En analysant ce tableau, nous constatons que :
- Les molécules d’ADN natif (A et F) ne sont pas dupliquées.
- Les molécules précisément définies à leur extrémité 5’ uniquement (B et D) augmentent de manière linéaire (de 1 par cycle sauf si l’ADN natif se dégrade).
- Le nombre de molécules simple ou double brin augmente selon une exponentielle d’ordre 2.
- Les molécules contenant la séquence exacte à amplifier (C et E) apparaissent dès le deuxième cycle et augmentent selon une suite arithmétique qui tend vers une exponentielle d’ordre 2 lorsque le nombre de cycles augmente. Notez que dès le dixième cycle, elles représentent près de 99% de l’ensemble.
- L’amplicon (les couples C-E) apparaît dès le troisième cycle et augmente selon une loi de même type que ses composants. Il représente près de 98 % des molécules au dixième cycle, 99,91 % au quinzième (zone où les ADNc issus d’ARNm fortement exprimés commencent généralement à devenir détectables).
Ces valeurs ont été obtenues en partant d’une seule molécule initiale d’ADN double brins, mais autrement, chaque matrice aurait subi le même processus. À un cycle donné, la quantité d’ADN dépend donc du nombre initial de matrices. En revanche, quelle que soit sa concentration initiale, il est théoriquement possible d’obtenir n’importe quelle quantité en ajustant le nombre de cycles. La PCR est donc régie théoriquement par la loi :
[ADN]cyclen = [ADN]initialexEn
Mais la PCR est une réaction enzymatique complexe. Le produit est identique au substrat et peut venir inhiber l’enzyme, mais surtout, les réactifs secondaires (amorces, dNTP) peuvent commencer à manquer. La PCR ne peut donc avoir une loi d’amplification exponentielle que tant que l’ADN matrice est le seul facteur limitant. La réaction devient ensuite imprédictible et il est alors impossible de pouvoir comparer plusieurs échantillons entre eux sans un biais quantitatif plus ou moins significatif (voir l’article sur la PCR quantitative). Il est donc important de bien comprendre les différentes phases d’une cinétique de PCR.