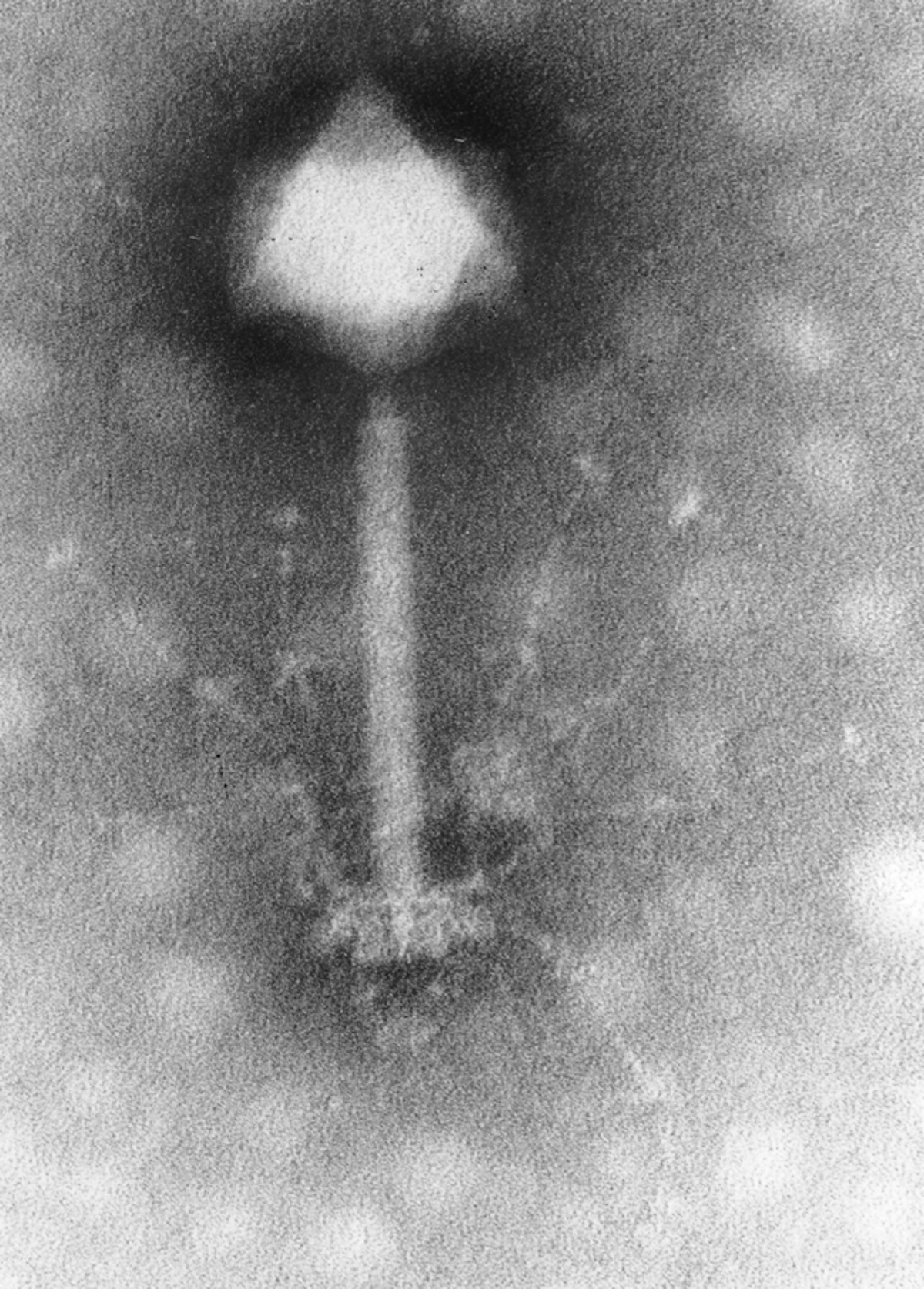
Bactériophage - Définition
La liste des auteurs de cet article est disponible ici.
Les bactériophages comme outil fondamental de recherche
Les phages ont permis l'essor de la biologie moléculaire
Dans les années 1960, des recherches de pointe menées sur les mécanismes hôte/phage par des physiologistes américains, Max Delbrück, Alfred Hershey et Salvador Luria, valurent à ces chercheurs le prix Nobel de médecine-physiologie en 1969.
Les phages ont permis différentes découvertes:
- L'expérience de Hershey et Chase qui a permis de confirmer la fonction de l'ADN en tant que support de l'information génétique. Hershey et Chase incorporèrent du phosphate 32 dans l'ADN d'une culture de phage et du soufre 35 dans les protéines d'une autre culture de ce même phage. Puis, ils utilisèrent chacune de ces cultures de phage indépendamment pour infecter E.Coli à raison d'un nombre élevé de particules virales par cellule bactérienne. Après un temps suffisant pour que l'infection ait eu lieu, ils détachèrent les enveloppes vides des phages (ghosts) des cellules bactériennes par agitation mécanique. Par centrifugation, ils séparèrent les cellules bactériennes des ghosts et mesurèrent la radioactivité des deux fractions obtenues. En utilisant les phages marqués au 32P, la majeure partie de la radioactivité aboutissait dans les cellules bactériennes, indiquant que l'ADN de phage entre dans les cellules. Au contraire, le 35S restait dans les ghosts montrant que les protéines du phage n'avaient jamais pénétré dans la cellule bactérienne. Conclusion: l'ADN est le matériel héréditaire tandis que les protéines de phages ne sont qu'un emballage qui est écarté une fois que le précieux ADN a été injecté dans la cellule bactérienne.
- En 1980, le biochimiste britannique Frederick Sanger reçut le prix Nobel pour avoir réussi à séquencer l'ADN en utilisant un phage. Le premier organisme biologique dont le génome a été séquencé est un phage ( intérêt étant dû à son ADN simple brin). Le protocole de la méthode de séquençage est le suivant : incubation de l'ADN à séquencer avec une amorce, le fragment de Klenow (ADN pol I dépourvue d'activité exonucléasique 5'→3'), les 4 désoxyribonucléotides (dNTP) et 1 didésoxyribonucléotide (ddNTP) en faible concentration. Le ddNTP induit l'arrêt de l'élongation: tous les fragments obtenus se termineront par ce nucléotide. Puisqu'il est utilisé en faible concentration, on va obtenir des fragments de tailles différentes. On refait l'expérience avec les 4 ddNTP. Les fragments sont séparés par électrophorèse sur gel de polyacrilamide.
- Les premières expériences suggérant un ARN intermédiaire dans la synthèse protéique. Il s'agit de l'expérience de E. Volkin et L. Astrachan en 1957. Ils font une expérience de pulse chase dans laquelle l'ARNm est marqué de façon spécifique avec de l'uracile radioactif. Ils constatent que l'infection par un bactériophage T2 induit une augmentation de la quantité d'ARNm dans la cellule hôte et que cet ARNm a un temps de vie très court (car très vite dégradé après le marquage). Conclusion: l'ARN joue un rôle intermédiaire entre l'ADN et les protéines.
- La découverte des enzymes de restriction en 1962 par W. Arber. Protocole de l'expérience: On infecte 2 souches bactériennes A et B par un phage X puis on récupère le lysat de phage pour l'utiliser dans une nouvelle infection dans les deux souches bactériennes utilisées précédemment. Résultat: le phage X-A peut infecter les 2 souches bactériennes alors le phage X-B ne peut pas infecter la souche A. Conclusion: les phages ont une spécificité d'hôte qui dépend de la souche dans lequel il s'est développé et pas de son génotype. Cette restriction d'hôte est due à la méthylation de l'ADN par des enzymes spécifiques permet de protéger l'ADN viral de la dégradation par des nucléases de la bactérie. Ces nucléases sont en fait des enzymes de restriction qui ne reconnaissent que la forme non méthylée de leur site de coupure. L'enzyme nécessaire à cette méthylation n'est présente que dans la souche A donc seul le phage X-A est méthylé donc seul lui n'est pas dégradé par les nucléases. Ce mécanisme permet a la bactérie de différencier son propre ADN de l'ADN étranger.
- La recherche en génétique sur la structure des génomes par Benzer. Celui-ci a déterminé la structure fine des gènes grâce à l'étude de recombinaisons entre mutants de bactériophage T4. Les bactériophages présentent deux avantages énormes : la fréquence de recombinaison est élevée, la descendance est quasi illimitée ce qui permettra d'avoir accès à des événements très rares.
L'étude des phages a des implications importantes en médecine et en génétique, en particulier pour la compréhension des infections virales, des anomalies génétiques, de l'embryologie humaine, des causes du cancer et de la résistance des bactéries aux antibiotiques.
Utilisation en génie génétique
Les phages sont utilisés de multiples manières dans la biologie moléculaire. Ils sont utilisés comme vecteurs de clonage pour insérer de l'ADN dans les bactéries. La méthode du phage display est une méthode qui permet la sélection d'un peptide grâce à sa présentation sur la surface de phages. Le phage display constitue une catégorie de phage permettant la construction de banques d'ADN ou d'ADN complémentaire. Les 2 principaux phages utilisés dans cette technique sont les phages M13 (phage filamenteux) et lambda qui infectent tous les 2 E.Coli. Prenons l'exemple du phage M13 qui est un phage filamenteux capable d'infecter uniquement les bactéries gram (-) ayant incorporé le facteur F et dont l'infection conduit à la lysogénie. Sa capside contient, entre autres, les protéines P8 et P3 nécessaire pour la liaison du bactériophage à la bactérie via les pilis sexuels. Ces 2 protéines vont être utilisé pour présenter à la surface des phages des molécules d'intérêt (peptide, fragment d'anticorps ou protéine entière): On va faire fusionner notre molécule d'intérêt avec les protéines P7 et P3, pour cela il faut insérer le gène codant la molécule d'intérêt à proximité de l'extrémité 5' des gènes P3 et P7 en respectant le cadre de lecture. On utilise l'une ou l'autre des protéines selon le type de molécule et la quantité de molécules qu'on veut exposer à la surface du phage. On distingue les phages polyvalent/homogène, où toutes les protéines P3 et P7 sont fusionnées, des phages monovalents/hétérogène ou seulement une partie des protéines le sont. Intérêt de la technique: elle permet d'obtenir des banques d'ADN qu'on peut facilement conserver et les clones sélectionnés sont multipliés à faible coût. Cette technique va permettre de produire des anticorps sans devoir passer par l'immunisation d'un animal. Limite de la technique: certaines molécules ne peuvent pas être exprimées comme par exemple les molécules toxiques pour la cellule hôte. Il y a donc une capacité limiter à transformer E.Coli.
Utilisation dans le séquençage de génomes entiers
Le séquençage d'un génome ne se fait pas d'un seul coup, mais petit à petit sur des fragments de génomes. Pour cela ces fragments d'ADN doivent être stockés et multipliés dans des organismes servant de banque d'ADN. Les phages en tant que vecteurs de clonage le permettent.