Acide ribonucléique messager - Définition
La liste des auteurs de cet article est disponible ici.
Synthèse et maturation
Transcription
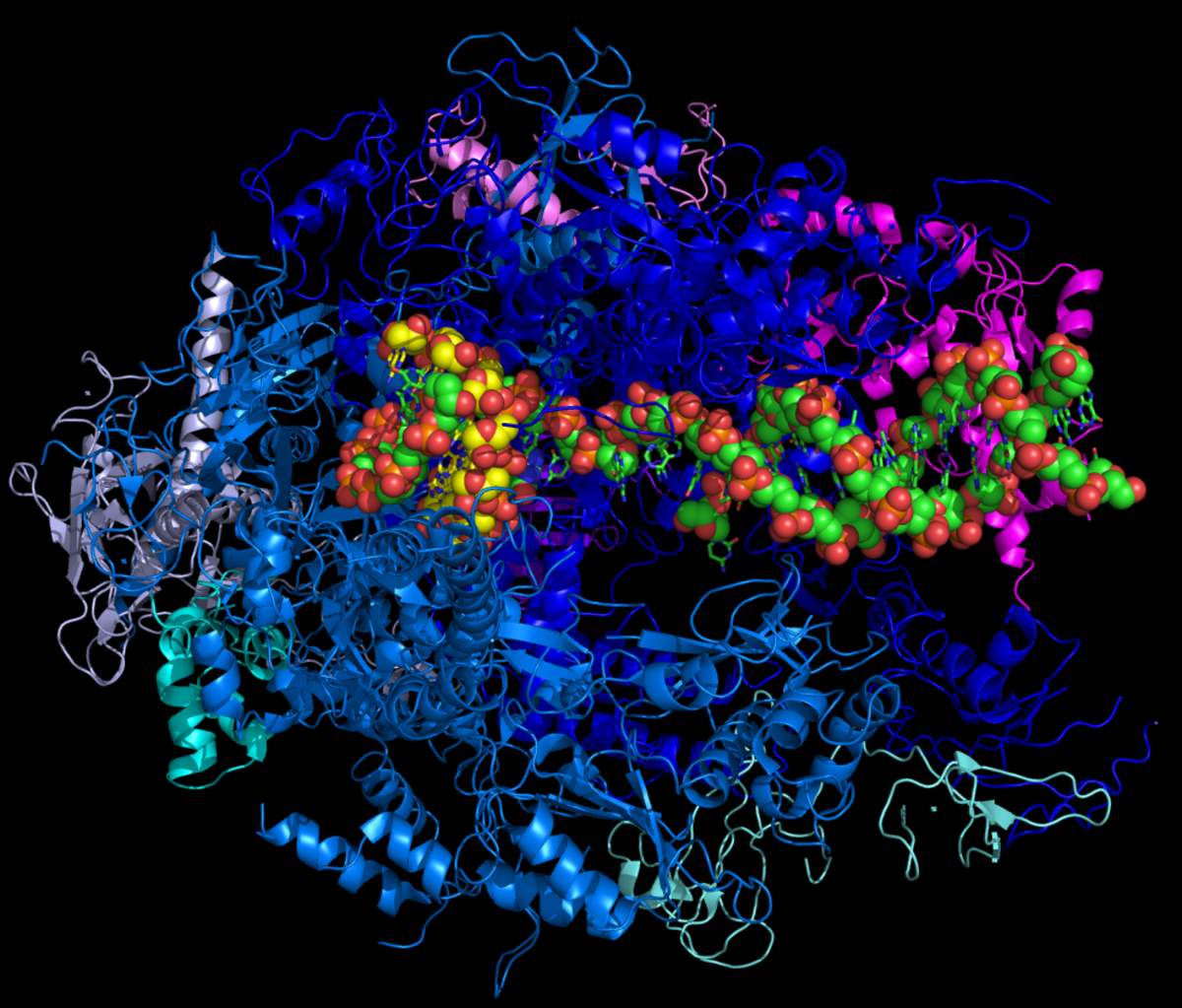
L'ARNm est en une copie locale d'une région de l'ADN correspondant à un ou quelques gènes codant des protéines. L'opération de copie appelée transcription se déroule dans le noyau de la Cellule chez les eucaryotes et dans le cytoplasme chez les procaryotes, c'est à dire là où réside le matériel génétique. La transcription a aussi lieu dans les organites semi-autonomes comme les mitochondries et les chloroplastes qui possèdent un génome indépendant. Cette transcription est effectuée par des enzymes spécifiques appelées ARN polymérases qui utilisent des ribonucléotides triphosphate comme monomères pour la synthèse de l'ARN. Chez les eucaryotes qui possèdent trois ARN polymérases distinctes dans leur noyau, c'est l'ARN polymérase II qui transcrit tous les ARNm cytoplasmiques, les deux autres ARN polymérases assurant la synthèse d'ARN stables non codants (ARN ribosomique, ARN de transfert...).
L' ARN polymérase se fixe sur une séquence spécifique de l'ADN appelée promoteur de transcription, juste en amont du début de l'ARNm. Elle sépare alors les deux brins du duplexe d'ADN et crée ce qu'on appelle une bulle de transcription puis synthétise la molécule d'ARN en se servant du brin d'ADN dit "brin transcrit" comme matrice. Un signal de terminaison, situé en aval du ou des gènes transcrits déclenche l'arrêt de la transcription, le détachement de l'ARN polymérase et la libération de l'ARNm terminé.
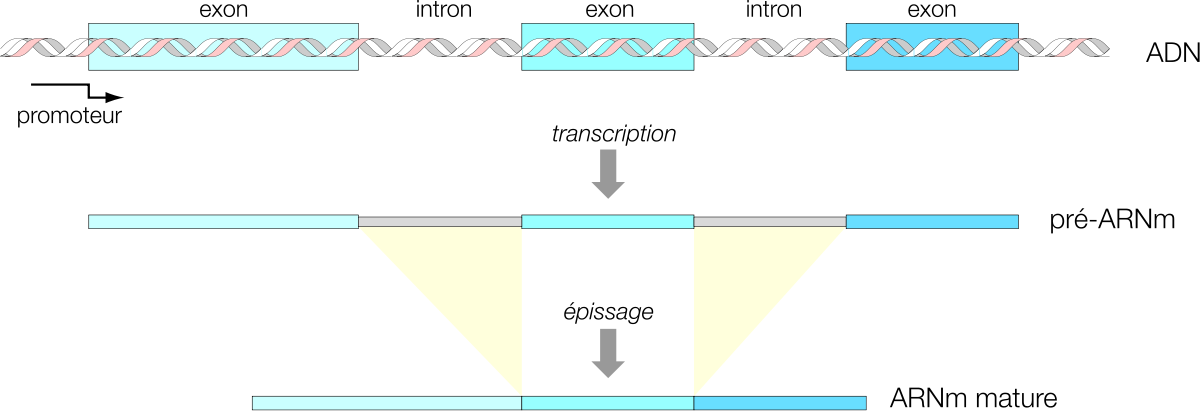
Chez les procaryotes, la molécule d'ARNm est le plus souvent directement traduite en protéines. Chez les eucaryotes (noyau et organites), la molécule synthétisée par les ARN polymérases est dans de nombreux cas de l'ARN pré-messager qui doit subir des maturations post-transcriptionnelles dans le noyau avant de pouvoir être traduit. En particulier, les parties codantes chez les eucaryotes sont en général interrompues par des séquences non codantes ou introns qui séparent les exons codants. Au cours de la maturation post-transcriptionnelle aura notamment lieu le phénomène d'épissage assurant la suture des séquences résultant de la transcription des exons alors que les séquences résultant de la transcription des introns seront retirées de l'ARN pré-messager.
Modifications post-transcriptionnelles
Chez les eucaryotes, après transcription de l'ADN, on obtient une molécule d'ARN précurseur ou ARN pré-messager qui n'est pas encore "mature", et qui ne peut pas, dans la majorité des cas, être directement traduite en protéine. Pour transformer ce pré-ARNm en ARNm mature, compétent pour la traduction, il faut lui faire subir deux types de modifications. Il faut modifier ses extrémités 5' (la coiffe) et 3' (queue poly (A)) afin en particulier d'augmenter sa stabilité mais aussi d'en permettre l'adressage vers la machinerie cellulaire adéquate, c'est à dire le ribosome dans le cytoplasme. Il faut aussi réaliser une étape complexe appelée épissage. Le pré-ARNm contient en effet dans sa structure une alternance d'introns et d'exons, dont seuls les exons sont codants pour la synthèse de la protéine. Les introns doivent donc être épissés (retirés) avant que le processus de traduction puisse s'effectuer. A l'issue de ces étapes qui se déroulent dans le noyau, on obtient un ARNm qui est alors exporté dans le cytoplasme.
Plusieurs facteurs interviennent dans ce processus. Après la transcription, le pré-ARNm s'associe à des protéines nucléaires pour former des ribonucléoprotéines nucléaires hétérogènes (hnRNP) qui interviennent dans la maturation et l'export. Plusieurs enzymes de modification du pre-ARNm sont directement associés à l'ARN polymérase II, ce qui permet d'agir sur le pré-ARNm au cours même de sa synthèse. On parle alors de modifications co-transcriptionnelles. Enfin, le processus d'épissage (voir plus bas) fait intervenir des petits ARN nucléaires (ARNsn) au sein de complexes snRNP. Ces complexes s'associent et se dissocient de manière ordonnée aux jonctions intron-exon pour en réaliser la coupure et la suture.
Modifications en 5'
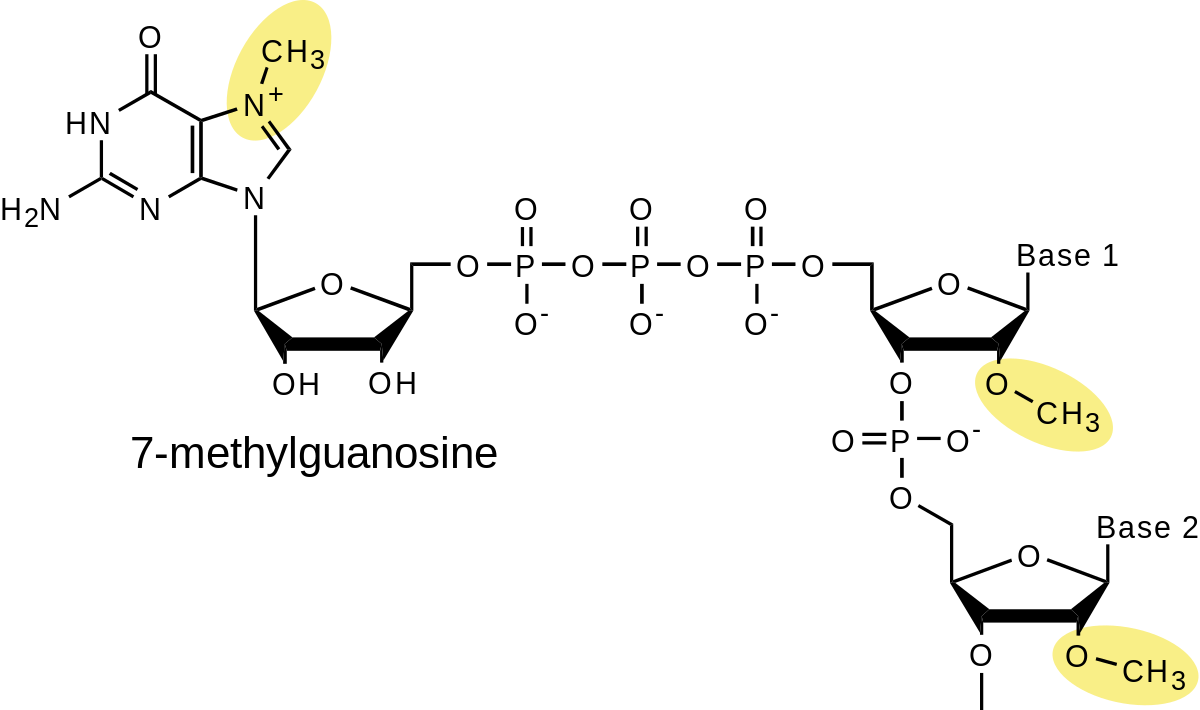
Chez les eucaryotes, l'extrémité 5' de l'ARNm subit une série d'étape post-transcriptionnelles successives qui vont aboutir à la formation de ce qu'on appelle la coiffe. Le rôle de cette coiffe est multiple, elle permet de stabiliser l'ARNm en le protégeant contre l'action de ribonucléases. La coiffe intervient également dans l'export de l'ARNm dans le cytoplasme (voir plus bas) et dans le recrutement du ribosome pour la traduction.
La formation de la coiffe est co-transcriptionnelle, elle se produit très rapidement après le début de la synthèse par l'ARN polymérase II, lorsque l'ARNm ne comporte qu'environ une trentaine de nucléotides. Ceci résulte du fait que ces modifications sont catalysées par des enzymes qui sont associées à l'ARN polymérase. Le synoptique des évènements est le suivant :
La première étape est le clivage de l'extrémité 5'-triphosphate de l'ARN précurseur par une triphosphatase. La triphosphatase laisse une extrémité 5' diphosphate. Il y a ensuite ajout d'une guanosine triphosphate par une guanylyl-transférase. Cette enzyme forme un pont 5'-5' triphosphate dans lequel la guanosine additionnelle est orientée tête-bêche. Il y a ensuite une série de méthylations, sur la position N7 de de la guanosine ajoutée et sur les 2'-hydroxyles des deux premiers riboses. Enfin, lorsque le premier nucléotide transcrit est une adénosine, on peut enfin avoir la formation de N6-méthyl-adénosine, par l'action d'une ARN méthylase qui est toutefois différente de l'ADN méthylase de maintenance.
L'ensemble de ces modifications a pour but de faire barrière à l'action des 5'-exonucléases, donc de protéger l'ARN de son environnement, mais joue aussi un rôle dans l'épissage, dans le transport nucléo-cytoplasmique et dans la traduction de l'ARNm en protéines.
Terminaison et modification en 3'
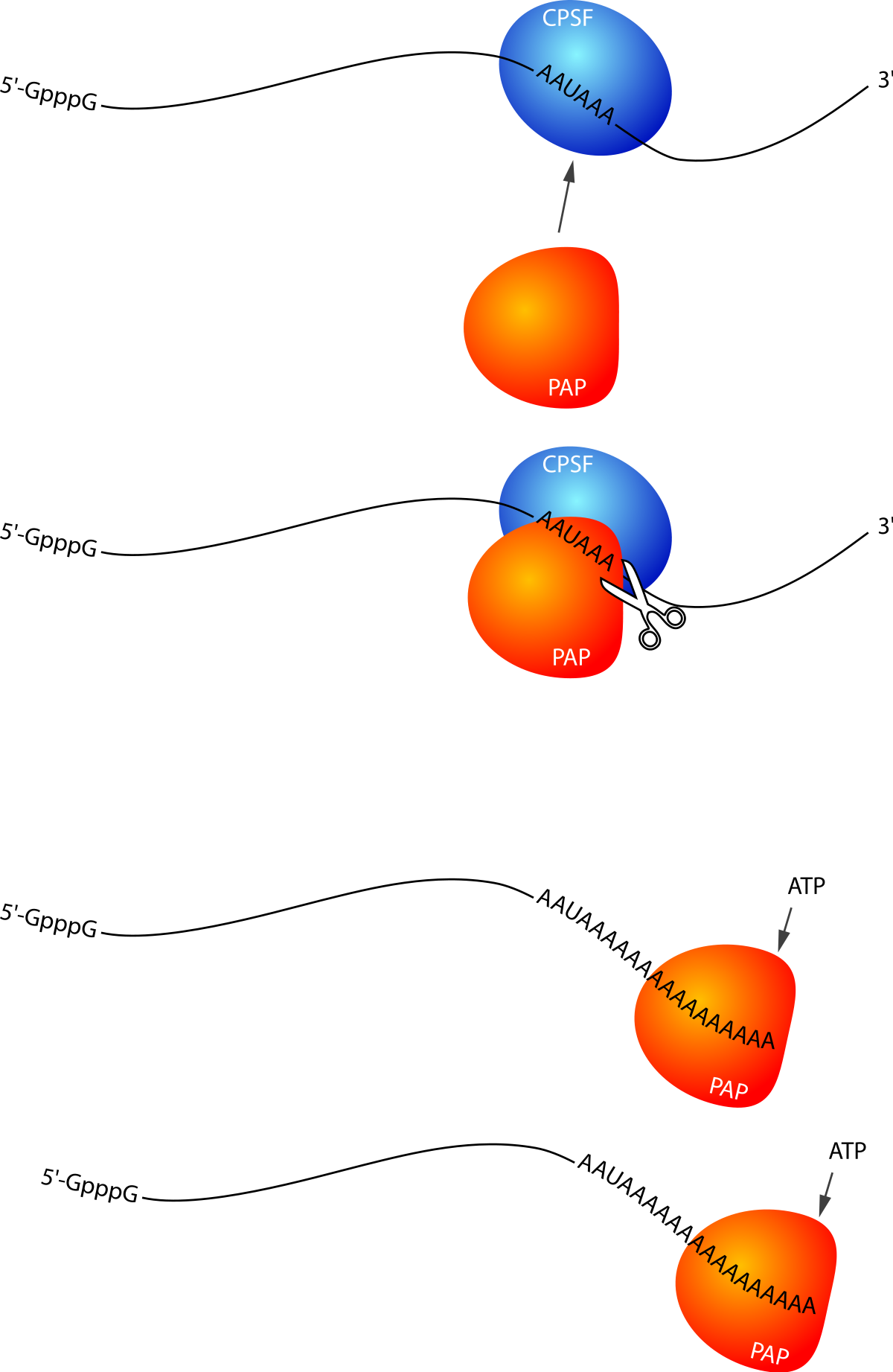
Chez les eucaryotes, la terminaison de la transcription se produit lorsque l'ARN polymérase atteint le signal de polyadénylation, 5'-AAUAAA-3', dans la région 3'-UTR de l'ARNm. Il y a alors recrutement d'un complexe protéique de coupure, CPSF (Cleavage and polyadenylation specificity factor). Le rôle de ce facteur est double, en interagissant avec l'ARN polymérase, il va provoquer l'arrêt de la transcription et le détachement de cette dernière. Ensuite il va cliver l'ARN juste en aval du signal AAUAAA, au niveau d'une séquence de type 5'-YA-3', où Y est une base pyrimidique (U ou C).
Après la coupure, CPSF va recruter la poly(A) polymérase qui va synthétiser une queue poly (A) composée de 100 à 250 résidus adényliques. Cette queue poly (A) joue plusieurs rôles, via le recrutement de protéines spécifiques, les PABP (poly (A) binding proteins). La polyadénylation et la fixation des PABP sur l'ARNm sont importantes pour l'export nucléaire, la protection contre les ribonucléases mais aussi 3'-OH, mais surtout pour la traduction en protéine (voir plus bas).
Chez les bactéries, le processus de terminaison de la transcription est différent, il passe principalement par la formation d'une structure secondaire sur l'ARNm (le terminateur), qui provoque une pause de l'ARN polymérase et son détachement de l'ADN matrice. Les ARNm bactériens ne sont en général pas polyadénylés en 3'.
Épissage
Les gènes eucaryotes ont fréquemment une structure mosaïque où les régions codantes pour une protéine sont morcelées en plusieurs segments (exons) séparés par des régions non codantes appelées introns. La transcription du gène produit initialement un pré-ARNm où on a donc une alternance exon/intron. L'épissage est le processus de maturation se déroulant dans le nucléoplasme qui permet l'excision des introns et la suture des différents exons pour obtenir un ARNm mature dans lequel le cadre de lecture de la protéine a été reconstitué.
Les jonctions amont et aval de chaque intron se caractérisent par des séquences de nucléotides conservées, qui sont reconnues par la machinerie d'épissage. Il existe également une séquence conservée à l'intérieur de l'intron, appelée "boite de branchement" et qui contient une adénosine strictement conservée, essentielle au mécanisme.
L'épissage est réalisé par une série de cinq particules ribonucléoprotéiques, les snRNP, chacune composée d'un ARN et de plusieurs protéines. Collectivement, ces cinq snRNP qui ont pour noms U1, U2, U4, U5 et U6, respectivement, sont appelés le spliceosome. Elles interviennent en séquence et se lient au pré-ARNm par le biais d'appariements de bases de type Watson-Crick.

Mécanisme de l'épissage
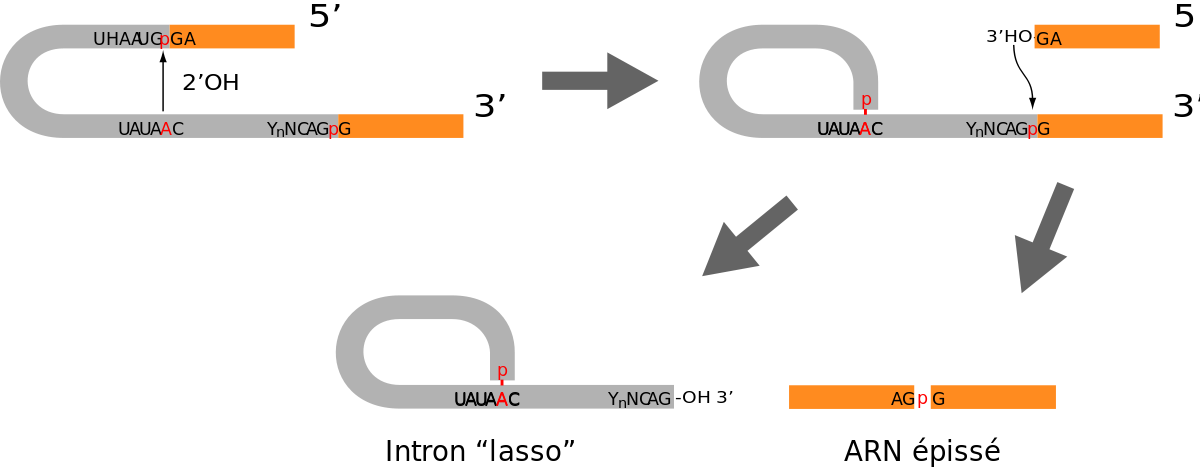
La première étape de l'épissage est un attaque de la liaison phosphodiester à la jonction entre le côté 5' de l'intron et l'exon amont. C'est le 2'-OH du ribose de l'adénosine conservée dans la boite de branchement qui joue le rôle de nucléophile dans cette réaction qui conduit à la formation d'un intermédiaire cyclique appelé "lasso" ((en) lariat) par transestérification. Dans cet intermédiaire il y a formation d'une liaison 2',5' phosphodiester entre la guanosine conservée à l'extrémité de l'intron et l'adénosine. Cette dernière est ainsi impliquée dans trois liaisons phosphodiester, en 5' et 3' avec les nucléotides précédents et suivants du pré-ARNm, et en 2' avec l'extrémité 5' de l'intron.
Ce premier transfert libère une extrémité 3'-OH libre sur l'exon amont. Celle-ci attaque alors à son tour la jonction aval de l'intron par un mécanisme analogue de transestérification ce qui aboutit à la suture des deux exons. L'intron sous forme de lasso est libéré et sera ultérieurement ouvert par une enzyme de débranchement pour permettre son recyclage.
Les snRNP interviennent dans ce processus en s'appariant aux jonctions de l'intron et des exons et à la boite de branchement. Ils maintiennent les différents acteurs pendant les deux étapes de transestérification et assurent le positionnement correct des différents groupements -OH réactif. Le snRNP U1 se fixe initialement sur la jonction exon/intron amont et le snRNP U2 sur la boite de branchement, U5 maintient les deux bords des deux exons et U6 joue probablement un rôle catalytique.
Edition
L'édition est un phénomène biologique qui permet à la cellule de modifier la séquence de l'ARN messager après la transcription. La séquence polypeptidique qui résultera de la traduction de cet ARNm ne correspond donc pas à la séquence exacte du gène correspondant. C'est une forme de régulation post-transcriptionnelle, ou de maturation de l'ARN. Ce processus, découvert initialement chez les mitochondries de trypanosomes, implique des facteurs protéiques et parfois des ARN guides. Il conduit soit à l'incorporation de nucléotides additionnels qui sont insérés dans l'ARNm, soit à la conversion chimique de certaines bases, par exemple du fait de l'action d'enzymes comme les cytidines ou les adénosines désaminases.