
Illustrationsbild Pixabay
Die Stabilität des genetischen Materials ist essenziell für das reibungslose Funktionieren jeder einzelnen unserer Zellen. Dennoch können Unfälle geschehen, zum Beispiel unter dem Einfluss von Strahlung oder durch Fehler bei der DNA-Replikation. Um diese Brüche zu reparieren, gibt es äußerst konservierte Mechanismen bei allen Arten, die für eine treue Reparatur verantwortlich sind.
Die homologe Rekombination ist ein hochpräziser Reparaturmechanismus für Brüche, der eine intakte DNA-Matrize mit identischer Sequenz nutzt. Diese Sequenz kann entweder von der Schwesterchromatide, vom weiter entfernten homologen Chromosom oder von anderen im Genom wiederholten Sequenzen stammen. Diese intakte homologe Kopie muss daher in der Weite des Genoms und des Zellkerns gefunden werden. Wie wird diese Suche nach einer homologen Nadel im Heuhaufen des Genoms durchgeführt?
Um diese Frage zu beantworten, haben Wissenschaftler des CNRS eine Hochdurchsatz-Genomtechnik namens ssHi-C bei der Hefe Saccharomyces cerevisiae entwickelt, einem Modellorganismus zur Untersuchung grundlegender DNA-Erhaltungsmechanismen. Mit dieser Technik konnten sie auf genomweiter Ebene die Kontakte kartieren, die durch den Bruch hergestellt wurden. Diese Daten ermöglichten es, zu enthüllen, dass die Suche nach Homologie in zwei großen Phasen abläuft:
- Eine erste Phase, in der die Strukturierung des Genoms durch Cohesine die Suche lokal einschränkt. Cohesine sind Proteine, die die Kohäsion der Schwesterchromatiden sicherstellen, indem sie die DNA binden.
- Eine zweite Phase, in der die Suche sich von diesen Einschränkungen löst und entfernte genomische Orte untersucht. Diese Fernsuche wird durch die Entstehung von langen einzelsträngigen DNA-Molekülen an beiden Seiten des Bruchs und deren Versteifung durch die Bildung eines Filaments der Rekombinase Rad51 ermöglicht. Dieses Protein, das Rekombinationsereignisse katalysiert, funktioniert in diesem Fall wie eine Angelrute, die ihre aus langen einzelsträngigen DNA-Molekülen bestehende Leine weit in den Zellkern auswirft. Die beiden Enden des Bruchs suchen koordiniert, und wenn ein Ende eine homologe Sequenz findet, intensiviert das andere Ende seine Suche in der Umgebung. Erstaunlicherweise binden einige Regionen des Genoms häufiger an den Bruch als andere, wie das Chromosom III. Das Genom wird also bei der Homologiesuche nicht gleichmäßig durchsucht, was die Existenz von Zielmechanismen nahelegt, die in zukünftigen Studien untersucht werden sollen.
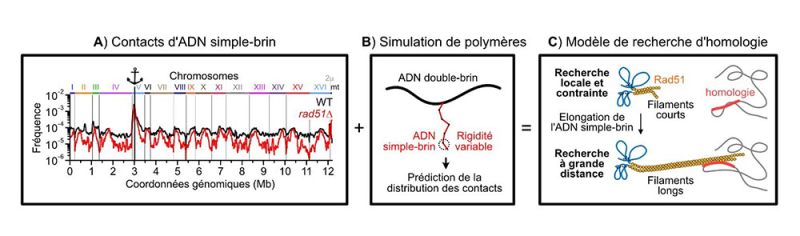
(A) Die ssHi-C-Technik ermöglicht es, die Kontaktfrequenz zwischen der bei einem Bruch generierten einzelsträngigen DNA (dargestellt durch den Anker) und dem Rest des Genoms in einem wilden oder mutierten Kontext zu bestimmen.
(B) Diese experimentellen Daten werden mit den Vorhersagen von Polymersimulationen verglichen.
(C) Dies führt zu einem Modell der Expansion der Homologiesuche, das auf der Bildung langer, steifer Filamente beiderseits des Bruchs durch das Rad51-Protein basiert.
© Aurèle Piazza
Diese Entdeckungen führen zu einem allgemeinen Modell der Homologiesuche, das auf der starren Struktur des Rad51-Filaments und seinen Partnern basiert. Es eröffnet neue Forschungsrichtungen, insbesondere um die durch Mutationen in Tumorsuppressorgenen wie den Rad51-Paralogen verursachten Defekte zu verstehen, die die Regulierung der Struktur des Rad51-Filaments und die Reichweite der Homologiesuche betreffen. Schließlich sollte die universell anwendbare ssHi-C-Technik es ermöglichen, die räumliche Lokalisierung der DNA während der Replikation oder ihrer Reparatur in vielen Organismen und zellulären Kontexten zu untersuchen.
Referenzen:
Mechanism of homology search expansion during recombinational DNA break repair in Saccharomyces cerevisiae.
A. Dumont, N. Mendiboure, J. Savocco, L. Anani, P. Moreau, A. Thierry, L. Modolo, D. Jost, A. Piazza.
Molecular Cell, 22. August 2024. DOI: 10.1016/j.molcel.2024.08.003