HIV, das AIDS-Virus, ist ein Retrovirus.
Im Elektronenmikroskop sind mit dem menschlichen Immunschwächevirus (grün-gelb) infizierte T-Lymphozyten zu sehen.
Bildnachweis: NIAID
Das Gag-Protein ist für die Assemblierung von Retroviruspartikeln unerlässlich...
Das Gag-Protein spielt eine zentrale Rolle bei der Assemblierung von Retroviren.
Einerseits ist es der Vorläufer der Strukturproteine der Viruspartikel: Ungefähr 2.000 Kopien von Gag setzen sich zusammen, um die unreifen Retroviruspartikel zu bilden; nach dem Ausknospen werden die Gag-Moleküle gespalten, und die Reifungsprodukte bilden die Matrix, das Kapsid und die Nukleokapsid der reifen, infektiösen Viruspartikel.
Andererseits wählt Gag die genomische RNA aus der Vielzahl zellulärer oder viraler RNA-Typen im Zytoplasma infizierter Zellen aus, um sie für die Einkapselung vorzubereiten. Dazu bindet sich Gag an eine Region der genomischen RNA, die je nach Retrovirus etwa 100 bis 300 Nukleotide lang ist und Signale zur Einkapselung enthält. Diese Region wird daher Psi (Packaging Signal) genannt. Psi zeichnet sich bei allen Retroviren durch das Vorhandensein von langreichweitigen Wechselwirkungen (LRI: Long Range Interactions) aus, die die globale Struktur von Psi aufrechterhalten.
... aber seine Bindung an das Genom reicht nicht aus, um die Einkapselung zu ermöglichen
Es war gut belegt, dass die Bindung von Gag an Psi notwendig für die selektive Einkapselung des Genoms von Retroviren ist, aber es war unklar, ob dies allein ausreicht, um diesen Vorgang zu fördern. Zudem war die Rolle der langreichweitigen Wechselwirkungen in diesem Prozess wenig bekannt. Um diese Fragen zu klären, verwendeten die Wissenschaftler das murine Mammatumorvirus (MMTV) als Modell.
Sie identifizierten Mutationen in einer neuen langreichweitigen Wechselwirkung, die die Einkapselung der genomischen RNA reduzieren, ohne die Affinität von Gag für Psi zu beeinträchtigen. Damit konnten sie zeigen, dass die Bindung von Gag an Psi notwendig, aber nicht ausreichend für eine effektive Integration der genomischen RNA in Retroviren ist.
Diese Mutationen beeinflussen die Gesamtstruktur von Psi, ohne jedoch die lokale Struktur der primären Bindungsstellen von Gag, die zuvor im Wildtyp-Virus von denselben Wissenschaftlern identifiziert wurden, zu verändern. Dennoch induzieren sie die Bindung von Gag an andere Regionen von Psi in den mutierten Viren.
Die Ergebnisse deuten darauf hin, dass die dreidimensionale Struktur des Komplexes, der zwischen Gag und Psi gebildet wird, die Assemblierung der Viruspartikel um die genomische RNA reguliert und somit die Einkapselung anderer viraler und zellulärer RNAs, die Gag mit hoher Affinität binden, verhindert.
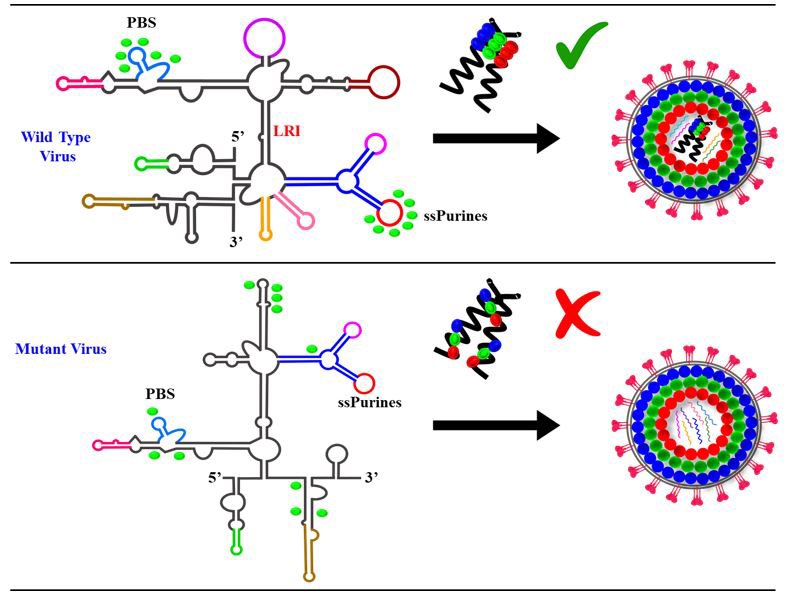
© Roland Marquet und Tahir Rizvi
Die Einkapselungssignale der genomischen RNA von MMTV enthalten eine in dieser Studie identifizierte langreichweitige Wechselwirkung (obere Abbildungshälfte). Im Wildtyp-Virus binden die Gag-Proteine (grüne Ovale) an die Bereiche PBS und ssPurine, was zur Einkapselung der genomischen RNA führt. Mutationen, die die langreichweitige Wechselwirkung zerstören (untere Abbildungshälfte), lösen die Bindung von Gag an andere Bereiche aus, ohne dabei die Affinität zu verlieren. Diese Bindung ermöglicht jedoch nicht die Integration der genomischen RNA in die Virusteilchen.
Referenzen:
MMTV RNA packaging requires an extended long-range interaction for productive Gag binding to packaging signals.
Suresha G. Prabhu, Vineeta N. Pillai, Lizna Mohamed Ali, Valérie Vivet-Boudou, Akhil Chameettachal, Serena Bernacchi, Farah Mustafa, Roland Marquet, Tahir A. Rizvi. Veröffentlicht: 3. Oktober 2024. Plos Biology. https://doi.org/10.1371/journal.pbio.3002827