Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)
En recherche, l'apprentissage automatique est de plus en plus utilisé pour détecter des tendances dans les ensembles de données complexes.
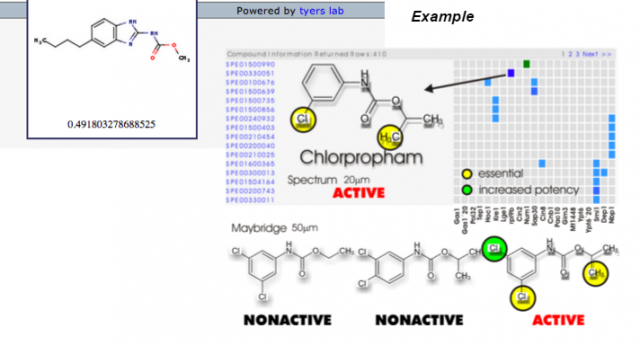
La découverte des médicaments de l'avenir pourrait reposer sur le travail des ordinateurs. Comme le démontre une étude de validation de principe publiée dans Cell Systems le 23 décembre dernier (1), un ordinateur disposant de données suffisantes sur les levures infectieuses peut apprendre à trouver des combinaisons de composés déjà connus ou jusqu'alors inconnus pouvant agir en synergie comme agents antifongiques. Bien qu'elle reste à perfectionner, cette méthode représente une nouvelle approche dans la lutte contre les maladies infectieuses, en permettant potentiellement de trouver rapidement les combinaisons d'agents qui contribueraient à vaincre la résistance aux médicaments.
Nous ignorons toujours dans quelle mesure les combinaisons de composés chimiques nouvellement découvertes agiront dans le traitement des infections aux levures chez les animaux ou les êtres humains. Toutefois, l'équipe de chercheurs a retenu 18 combinaisons potentielles pour traiter les infections aux levures pathogènes chez l'humain dans des récipients de laboratoire. Leur taux de réussite élevé confirme que ces combinaisons pourraient donner lieu à la mise au point de médicaments. Certaines des combinaisons se sont notamment avérées inoffensives pour les lignées cellulaires humaines.
"Notre étude montre qu'il est possible d'utiliser des modèles relativement simples, mais constitués d'éléments extrêmement complexes, comme les levures, pour mieux comprendre comment les composés chimiques et les médicaments interagissent avec des systèmes biologiques, indique Mike Tyers, professeur de biologie des systèmes à l'Université de Montréal et auteur principal de l'étude. Ces concepts pourront certainement être appliqués à des problèmes plus épineux en santé humaine."
Des ordinateurs qui apprennent
Dans plusieurs domaines de recherche, les équipes utilisent maintenant l'apprentissage automatique pour détecter des tendances dans les ensembles de données complexes, par exemple pour reconnaître des images sur le Web ou dans des systèmes de commande robotique."Cette façon de faire s'est accentuée récemment dans le secteur des sciences biologiques, où les chercheurs mettent de plus en plus à profit l'apprentissage automatique pour interpréter les énormes ensembles de données à l'échelle du génome, souligne Jan Wildenhain, développeur de systèmes à l'Université d'Édimbourg et l'un des premiers auteurs de l'étude. Les données biologiques sont devenues tout simplement trop nombreuses pour que nous puissions les traiter en nous en remettant seulement à l'intuition humaine."
Pour leur premier algorithme d'apprentissage automatique, les chercheurs ont utilisé la levure de bière (S. cerevisiae), car il s'agit de la seule levure dont le réseau génétique est connu. Ainsi, bien que les bactéries résistantes aux médicaments représentent actuellement le principal enjeu de santé publique, le modèle des levures offre un ensemble de données beaucoup plus grand et informatif pour ce type d'étude.
Les chercheurs ont commencé par entrer dans l'ordinateur les données génétiques tirées des décennies de recherche sur les levures (soit un ensemble de 195 souches génétiquement différentes) et les réponses génétiques de ces souches soumises à des écrans chimiques (un ensemble diversifié de 4915 composés a été utilisé), de sorte que l'ordinateur puisse concevoir des modèles à partir des interactions entre les composés chimiques et les gènes. Les données n'étaient toutefois pas suffisantes, et l'algorithme initial avait un faible pouvoir prédictif.
"À cette étape du projet, nous étions extrêmement déçus et nous sommes retournés à la planche à dessin, affirme Michaela Spitzer, boursière de recherche postdoctorale à l'Université McMaster et aussi première auteure de l'étude. Nous savions que les structures chimiques et le réseau génétique des cellules devaient jouer un rôle dans les effets synergiques chimiques que nous détections expérimentalement. Cependant, nous ne savions pas clairement comment procéder à la déconvolution de ces liens à partir de centaines de milliers de points de données. Finalement, nous avons dû revoir nos modèles plusieurs fois en fonction des ensembles de données servant à la préparation des algorithmes, puis mettre à l'essai les modèles dans différentes banques de composés que le modèle n'avait jamais obtenus auparavant."
La base de données ChemGRID, une ressource
Les algorithmes ont été préparés avec un ensemble de 1221 composés uniques ayant servi à créer et à tester 8128 combinaisons. Toutes les données de l'étude peuvent être téléchargées et examinées sans restriction à partir de la base de données ChemGRID."Nous espérons que d'autres groupes testeront nos modèles, tout comme nous continuerons très certainement de le faire. Peut-être même que quelqu'un en viendra à créer de meilleurs modèles, souhaitent Michaela Spitzer et Jan Wildenhain. Il serait très intéressant d'appliquer notre approche d'apprentissage automatique à des ensembles de données totalement différents pour prédire les synergies."
En plus de collaborer avec d'autres laboratoires, le groupe prévoit mettre en place une approche d'apprentissage automatique semblable pour se pencher sur les interactions entre les composés chimiques et les gènes dans les cellules humaines à l'aide de la technologie d'édition génique CRISPR/Cas9. En recueillant ces données, l'équipe pourrait créer un algorithme qui prédirait les combinaisons de composés chimiques capables de distinguer les cellules saines de celles qui sont malades, comme les cellules cancéreuses.
Ces travaux ont été rendus possibles grâce au soutien des chaires de recherche du Canada en études moléculaires des antibiotiques, et en biologie des systèmes et biologie synthétique, à la subvention International Research Scholar du Howard Hughes Medical Institute, à la subvention Royal Society Wolfson Research Merit Award, au soutien de la Scottish Universities Life Sciences Alliance Research Chair, à des bourses des Instituts de recherche en santé du Canada, du Conseil européen de la recherche, du Wellcome Trust, des National Institutes of Health, à une subvention du ministère de l'Éducation, de l'Enseignement supérieur et de la Recherche du Québec par l'entremise de Génome Québec et à l'appui de l'Office of the Assistant Secretary of Defense for Health Affairs des États-Unis par l'entremise du Breast Cancer Research Program.
Note:
(1). J. Wildenhain et autres, "Prediction of Synergism from Chemical-Genetic Interactions by Machine Learning",Cell Systems,2015.
Populaires