Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)
Préserver les humains des expositions au mercure est le principal objectif de la Convention de Minamata des Nations Unies ratifiée en août 2017. Or, si cette convention pointe vers la nécessité de réduire les émissions anthropiques de mercure inorganique, elle ne prend pas en compte la forme organique du mercure, le méthylmercure, qui est sa forme la plus toxique et surtout la seule capable de s'amplifier le long de la chaîne trophique et ainsi de mettre en péril les écosystèmes marins et la santé humaine. La consommation de poissons marins est en effet la principale voie d'exposition de l'homme au mercure.
Le méthylmercure est produit dans l'océan par des microorganismes à partir de mercure inorganique (1). Cette méthylation du mercure inorganique marin a été confirmée dans tous les bassins océaniques, quels que soient les niveaux d'oxygénation de leurs eaux. Pourtant, jusqu'en 2016, seuls les microorganismes anaérobies (2) étaient connus pour produire du méthylmercure. C'était le paradoxe de l'origine du méthylmercure.
En 2016, il a été démontré que la bactérie microaérophile (2) Nitrospina détectée dans la banquise antarctique possédait les gènes de la méthylation du mercure. Il a également été suggéré qu'elle pourrait aussi être responsable de la production de méthylmercure dans d'autres environnements oxiques (pourvus en oxygène) sans néanmoins que cela ait pu être confirmé par les métagénomes océaniques disponibles. La question de savoir "qui méthyle le mercure dans l'océan global" n'était donc toujours pas résolue. Résoudre cette énigme est pourtant d'une importance capitale pour mieux comprendre les liens entre les émissions anthropiques de mercure et le transfert du méthylmercure le long des chaînes trophiques.
De jeunes chercheurs du MIO ont combiné leurs expertises en bioinformatique, microbiologie et biogéochimie marine afin d'appréhender ce problème. Sur la base de jeux exhaustifs de données métagénomiques (3) et métatranscriptomiques3 issus du projet TARA OCEANS (www.oceans.taraexpeditions.org), ils ont pu identifier, dans tous les bassins océaniques couverts par l'expédition, des gènes de méthylation du mercure. Ces gènes sont non seulement présents, mais aussi transcrits (4), ce qui démontre le rôle actif des microorganismes identifiés dans la méthylation du mercure.
Les résultats obtenus ont permis d'identifié la bactérie microaérophile nitrite-oxidante Nitrospina comme la productrice prédominante et omniprésente de méthylmercure dans les eaux oxiques de subsurface de l'océan global. En revanche, les bactéries sulfato-réductrices, déjà bien connues et considérées comme les principales productrices de méthylmercure dans d'autres écosystèmes comme les sédiments, représenteraient une fraction mineure des bactéries porteuses du gène de méthylation du mercure dans l'océan.
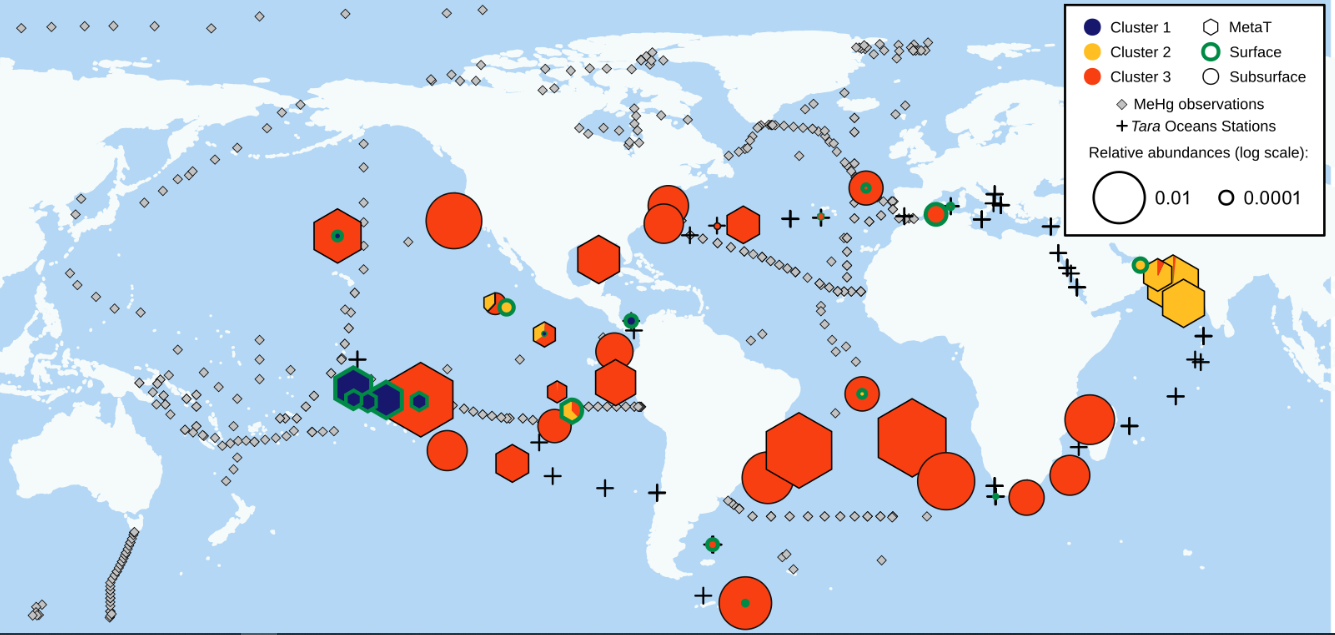
Les cercles colorés indiquent les stations TARA OCEANS dans lesquelles le gène responsable de la méthylation du mercure (hgcA) a été identifié, les hexagones colorés celles dans lesquelles le gène est non seulement présent mais aussi actif (54 % des cas) et les croix celles dans lesquelles le gène n'a pas été détecté. La couleur rouge des symboles indique l'affiliation du gène à la bactérie microaérobie Nitrospina prédominante et omniprésente dans les eaux oxiques de subsurface de l'océan global (les autres couleurs correspondant à d'autres bactéries).
Tout en résolvant le paradoxe apparent de l'origine du méthylmercure marin, ces travaux conduisent à une profonde reconsidération du rôle de Nitrospina dans la production de méthylmercure, rôle qui s'avère majeur non seulement dans la banquise antarctique mais aussi dans les océans Pacifique, Atlantique, Indien et Austral. Ce faisant, ils apportent une contribution importante à la compréhension du cycle global du mercure qui permettra de mieux estimer les effets du changement climatique sur la production marine de méthylmercure.
Référence publication:
Villar, E.; Cabrol, L.; Heimbürger‐Boavida, L.E. Widespread Microbial Mercury Methylation Genes in the Global Ocean. Environ. Microbiol. Rep. 2020, 1758-2229.12829.
https://sfamjournals.onlinelibrary.wiley.com/doi/abs/10.1111/1758-2229.12829
Notes:
(1) Les sources primaires du mercure fournissent l'essentiel du mercure inorganique, les sources primaires naturelles étant de nature volcanique ou hydrothermale.
(2) Les microorganismes aérobies ont besoin d'oxygène pour vivre. Pour les microorganismes anaérobies, 1'oxygène est toxique. Les microorganismes microaérophiles ont besoin d'un peu d'oxygène pour vivre, lequel devient toxique à des concentrations trop élevées.
(3) La métagénomique étudie l'ADN (les gènes) alors que la métatranscriptomique étudie l'ARN (les transcrits). La simple présence d'un gène ne suffit pas pour dire si la bactérie est active ou pas, c'est-à-dire si elle transforme effectivement son gène en protéine utile. En revanche, la transcription indique que les microorganismes sont bien métaboliquement actifs.
(4) La transcription en ARN est la première étape de l'expression d'un gène, c'est-à-dire du passage de l'information génétique stockée dans l'ADN à la fabrication de molécules fonctionnelles effectives et opérationnelles comme les enzymes et autres protéines.
Contacts:
- Lea Cabrol - MIO/IEB - lea.cabrol at mio.osupytheas.fr
- Lars-Éric Heimbürger-Boavida - MIO/PHYTHEAS - lars-eric.heimburgern at mio.osupytheas.fr